
44
n. 20 novembre 2015
AMBIENTALE
MONITORAGGIO
suggerendo interventi sanitari
adeguati.
Card-Fish
Per studio delle comunità
batteriche, tra le tecniche
di ibridazione in situ più
comunemente utilizzate, vi sono la
Fluorescence In-Situ Hybridization
(Fish) e la sua evoluzione
Catalyzed Reported Deposition-
Fish (Card-Fish). Queste tecniche
si basano sull’uso di sonde
molecolari oligonucleotidiche
rRNA specifiche, che si legano
a segmenti complementari del
corredo genetico e, mediante
la marcatura con un composto
fluorescente, rendono la cellula
individuabile mediante microscopia
ad epifluorescenza. La Card-Fish
prevede l’utilizzo di sonde rRNA
specifiche marcate con horseradish
peroxidase (HRP). Una singola
HRP catalizza la deposizione di
molta tirammide, marcata con un
fluorocromo, che si lega ai residui
di tirosina all’interno della cellula,
amplificando l’intensità del segnale
di fluorescenza. La Card-Fish
permette infatti di identificare
anche microrganismi caratterizzati
da una bassa attività metabolica
(cellule poco attive, bassa densità
dei ribosomi, es. la maggior parte
dei batteri in ambiente acquatico).
Inoltre, in alcuni campioni la
presenza di un forte segnale di
fondo potrebbe rendere difficile
la visualizzazione delle cellule.
Usando la Card-Fish la percentuale
di microrganismi identificati nei
sistemi marini è incrementata
di tre volte permettendo quindi
un’accurata quantificazione per
esempio negli oceani. Un punto
critico della tecnica è rappresentato
dalle grandi dimensioni della
sonda marcata con HRP (circa 44
kDa). Per questo motivo vengono
utilizzati uno o la combinazione di
più enzimi (lisozima-peptidoglicano,
proteasi-parete proteinacea)
con lo scopo di permeabilizzare
sufficientemente la parete senza
arrivare alla rottura della cellula
e permettere il passaggio delle
sonde.
Per identificare e quantificare le
clamidie in ambiente costiero
è stato ottimizzato il protocollo
della Card-Fish da Pizzetti
et
al.
[7]. Nello specifico è stato
necessario utilizzare il lisozima
insieme all’acromopeptidasi in
quanto la parete di questi batteri
è per lo più proteinacea e sono
state disegnate ed ottimizzate
due sonde specifiche, Chl282
e Chl282bis, che combinate
insieme hanno permesso la
quantificazione dell’intero ordine
delle Chlamydiales [7].
La densità batterica stimata
attraverso microscopia ad
epifluorescenza (Figura 2) è
espressa come numero di cellule
(cells) per millilitro (mL) nel caso di
campioni d’acqua ed è indicativa,
dell’abbondanza assoluta dei
microrganismi specifici che si
stanno quantificando, per esempio
le clamidie, ma anche della loro
relativa abbondanza rispetto al
numero totale di batteri presenti nel
campione ambientale.
Per uno studio approfondito
della relazione tra le clamidie
e l’ospite viene utilizzato il
microscopio confocale (Clsm)
che permette una ricostruzione
tridimensionale dell’immagine.
Spesso il Clsm viene combinato
con un sistema a super-risoluzione
includendo la tecnica Super-
resolution Structured Illumination
Fluorescence Microscopy (SR-
SIM) che permette, grazie alla
più alta risoluzione, sia di meglio
visualizzare che di quantificare
le clamidie all’interno dell’ospite
(Figura 3). È quindi possibile
identificare rapidamente non solo
clamidie a vita libera ma anche
quelle associate ad ospiti intermedi.
Il campionamento per l’analisi
Card-Fish è molto rapido e
prevede il prelievo di acqua
in quantità variabili in base al
campione da analizzare. I tempi
di analisi sono rapidi e, in seguito
ad una fase di fissaggio del
campione fresco in formaldeide
(essenziale per mantenere
l’integrità morfologica delle cellule
ed iniziare a permeabilizzare la
parete cellulare) e filtrazione su
appositi filtri è possibile eseguire
l’ibridazione e ottenere il dato
quantitativo finale entro le 24 h
successive. Il campione filtrato
si può conservare per diversi
mesi e può essere utilizzato per
successive analisi. Nello specifico,
le clamidie ambientali essendo
considerate ‘rare populations’
dovrebbero essere quantificate
su filtri contenenti elevati volumi
di campione per sopperire alla
limitazione nel quantificarle.
Isolamento di clamidie
ambientali
Recentemente, analisi molecolari
hanno mostrato un’alta diversità
delle clamidie, soprattutto in
ambiente marino [8], ma tale
diversità rimane sottostimata e
gli ospiti naturali sono, in molti
casi, sconosciuti. Per questo
motivo si ricorre a tecniche di
co-coltivazione con ospiti di
clamidie ambientali ben conosciuti
(per esempio Acanthamoeba
spp., Hartmannella spp.) per
isolare nuovi ceppi di clamidie
da diversi ambienti acquatici in
grado di sopravvivere e dividersi
all’interno dell’ospite potenziale.
In parallelo, tramite la tecnica a
diluizione fino ad estinzione, è
possibile isolare direttamente
l’ospite naturale ed identificarlo
tramite microscopia [ottica ed a
contrasto interferenziale (DIC)] ed
amplificazione e sequenziamento
del gene per il 18S rRNA. Una volta
isolato l’ospite, per determinare
la presenza delle clamidie al suo
interno viene utilizzata la Fish
tramite sonde oligonucleotidiche
specifiche per l’identificazione
delle clamidie (Figura 4). Ogni
clamidia isolata è successivamente
analizzata attraverso il
sequenziamento del gene per
il 16S rRNA ed il microscopio
elettronico a trasmissione (TEM).
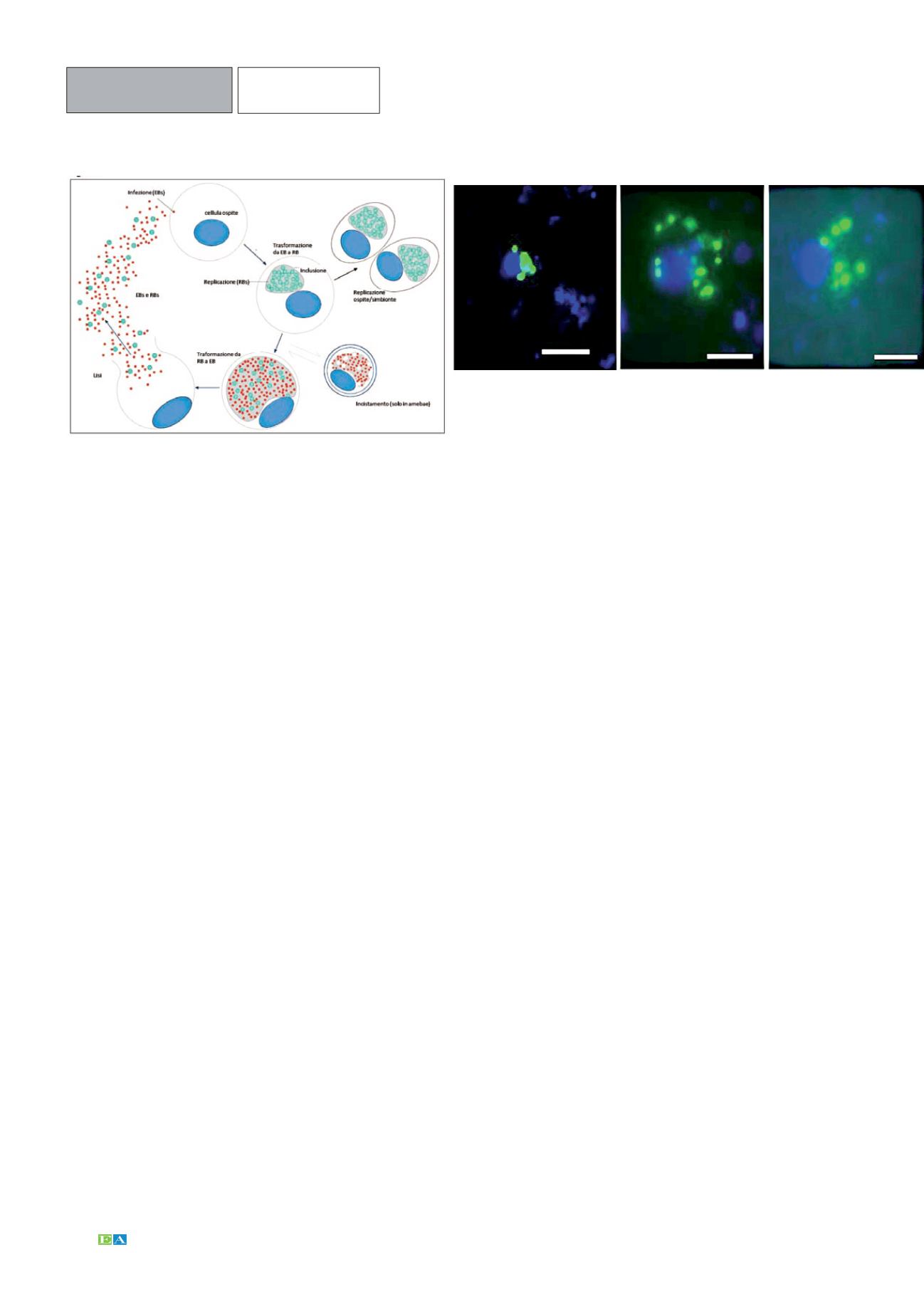
Figura 1 - Ciclo vitale delle Chlamydiae (modificato da [2]).
Figura 2 - Associazione protisti-clamidie - Immagine al microscopio ad
epifluorescenza: Blu, segnale DAPI che lega il DNA; verde, segnale fluoresceina di
cellule di Chlamydiae identificate tramite Card-Fish (sonda Chl282 combinata con la
Chl282bis). Scala: 5 µm.



















